Projekt auf einen Blick
Krebstherapien sind in den vergangenen Jahren durch zielgerichtete Therapien und Immuntherapien deutlich verbessert worden, was die Überlebenschancen erheblich erhöht, aber auch die Behandlung komplexer macht. Die Präzisionsonkologie – personalisierte Medizin auf Basis genetischer Tumorprofile – erfordert hochspezialisierte Therapieentscheidungen. Onkologische Leitlinien sollen durch evidenzbasierte Empfehlungen eine vergleichbare Behandlung aller Krebspatientinnen und -patienten sicherstellen. In der Praxis zeigt sich jedoch häufig eine Diskrepanz: Studien zeigen, dass weltweit nur teils etwa 50% der Patientinnen und Patienten leitliniengerecht behandelt werden.
In Kooperation mit dem Kantonsspital Graubünden hat sich die Projektgruppe das Ziel gesetzt, die Behandlung von onkologischen Patientinnen und Patienten zu verbessern und zu standardisieren. Dies soll durch eine datengetriebene Analyse erreicht werden, die auf den sogenannten Patienten- bzw. Behandlungspfaden als Datengrundlage basiert. Mithilfe einer strukturierten Modellierung und Visualisierung der onkologischen Behandlungspfade können Abweichungen zu den vorhandenen Leitlinien und damit Engpässe in der Versorgung identifiziert und durch den Einsatz von Methoden der künstlichen Intelligenz gezielt Lösungsansätze entwickelt werden. Langfristig trägt dies zu einer besseren Versorgungsqualität bei und kann gleichzeitig zu einer Reduktion oder Optimierung der Behandlungskosten durch Vereinheitlichung sowie zu einer nachhaltigen Entlastung des Gesundheitssystems führen.
Projekt
KI-Hilfe für Patientenpfade in der Onkologie erstellenLead
Institut für Data Analysis, Artificial Intelligence, Visualization und Simulation (DAViS) Mehr über Institut für Data Analysis, Artificial Intelligence, Visualization und Simulation (DAViS)Projektleitung
Staudt Yves Mehr über Staudt YvesBeteiligte
Kantonsspital GraubündenTeam
Marxer Curdin Mehr über Marxer Curdin Rölke Heiko Mehr über Rölke HeikoForschungsfelder
AI for structured and unstructured data Mehr über AI for structured and unstructured data Visualization and Dashboards Mehr über Visualization and DashboardsAuftrag/Finanzierung
Grundfinanzierung, Förderverein Fachhochschule GraubündenDauer
Januar 2024 – April 2025
Ausgangslage
Die stetig steigenden Gesundheitskosten, bedingt durch die Alterung der Bevölkerung, die Zunahme chronischer Erkrankungen und den Einsatz immer komplexerer medizinischer Behandlungen, stellen Gesundheitssysteme weltweit vor grosse Herausforderungen. Auch in der Schweiz setzt sich dieser Trend fort: Die Spitalausgaben nehmen kontinuierlich zu, während gleichzeitig der Anspruch an eine qualitativ hochwertige und effiziente Versorgung wächst.
Besonders deutlich zeigt sich die Komplexität in der Onkologie. Durch die Einführung zielgerichteter Therapien, Immuntherapien und der Präzisionsonkologie hat sich die Behandlung von Krebspatientinnen und -patienten stark weiterentwickelt. Diese Fortschritte erhöhen zwar die Überlebenschancen, führen jedoch zu aufwendigen Therapieentscheidungen, die eine präzise und strukturierte Planung erfordern. Obwohl klinische Leitlinien evidenzbasierte Empfehlungen für eine vergleichbare Versorgung geben, werden sie in der Praxis oft nur teilweise umgesetzt. Gründe dafür sind unter anderem institutionelle Unterschiede, variierende Behandlungsroutinen sowie die überwiegend unstrukturierte Dokumentation medizinischer Abläufe.
Diese fehlende Standardisierung erschwert die systematische Analyse von Behandlungspfaden, die Identifizierung von Abweichungen von Leitlinien und die Aufzeichnung von Optimierungspotenzialen. Gleichzeitig wächst der Bedarf an datengetriebenen Methoden, um die Versorgungsqualität zu verbessern und Ressourcen effizienter einzusetzen. Technologische Ansätze wie künstliche Intelligenz (KI), Natural Language Processing (NLP) und Process Mining bieten hier neue Möglichkeiten: Sie ermöglichen, unstrukturierte medizinische Texte in analysierbare Datenformate zu überführen, Behandlungsschritte transparent abzubilden und dadurch fundierte Entscheidungsgrundlagen für eine nachhaltige Optimierung des Gesundheitssystems zu schaffen.
Projektziel
Ziel dieses Projekts ist es, Patientenprozesse anhand klar definierter Behandlungsschritte strukturiert darzustellen und zu analysieren. Dadurch sollen onkologische Behandlungspfade nachvollziehbar abgebildet und systematisch ausgewertet werden können. Ein besonderer Fokus liegt dabei auf der Identifikation von Abweichungen gegenüber medizinischen Leitlinien sowie auf der Erkennung möglicher Versorgungsengpässe und Ineffizienzen.
Im Rahmen des Projekts wurde zunächst die Grundlage geschaffen, indem untersucht wurde, wie unstrukturierte medizinische Texte automatisiert in standardisierte Codesysteme wie ICD-10 oder CHOP überführt werden können. Diese automatisierte Kodierung ermöglicht eine einheitliche Datenbasis, auf der weiterführende Analysen aufbauen können. Anschliessend wurden erste Behandlungspfade mittels statistischer und prozessorientierter Methoden (Process Mining) rekonstruiert und visualisiert, um exemplarisch den tatsächlichen Verlauf onkologischer Behandlungen abzubilden.
Diese Ergebnisse stellen die Basis für weiterführende Entwicklungen dar. In kommenden Projektphasen sollen die erarbeiteten Analyseverfahren auf klinische Routinedaten übertragen und in eine interaktive Visualisierung integriert werden. Ziel ist es, medizinischem Fachpersonal ein intuitives Dashboard bereitzustellen, das die Interpretation von Behandlungspfaden erleichtert und datenbasierte Entscheidungen unterstützt.
Langfristig trägt dieses Projekt dazu bei, die onkologische Versorgung zu standardisieren, die Behandlungsqualität zu erhöhen und eine effizientere Nutzung der verfügbaren Ressourcen zu ermöglichen.
Umsetzung
Der Weg zu einer datengetriebenen Patientenpfadanalyse ist jedoch anspruchsvoll. Medizinische Behandlungen werden hauptsächlich in uneinheitlicher Textform dokumentiert. Dieses Format ist zwar für Menschen gut verständlich, erlaubt es Computern jedoch nur begrenzt, Behandlungspfade strukturiert zu modellieren. Daher wurde durch das Projektteam im ersten Schritt untersucht, wie medizinische Texte in strukturierte Datenformate überführt werden können.
In der medizinischen Praxis in der Schweiz, insbesondere mit dem Fokus auf dem Abrechnungswesen, werden Behandlungen und Diagnosen mittels standardisierter Codes dokumentiert, wie beispielsweise der Schweizerischen Operationsklassifikation (CHOP) oder der International Statistical Classification of Diseases and Related Health Problems, 10th Revision (ICD-10). Es existieren mehrere solcher Kodierungssysteme, die international verwendet werden. Einige davon decken nur spezifische Bereiche wie die ambulante, stationäre oder systemische Therapie ab, während andere einen ganzheitlicheren Ansatz verfolgen, wie etwa SNOMED CT (Systematized Nomenclature of Medicine – Clinical Terms).
Die Überführung von medizinischen Texten in solche Codierungssysteme wird in der Forschung unter dem Begriff automatische Codierungssysteme zusammengefasst. In einer internationalen Zusammenarbeit mit der University of San Francisco hat das Projektteam kleine Sprachmodelle trainiert, die diese automatische Codierung übernehmen können. Dabei fiel bewusst der Fokus auf kleine Sprachmodelle, da viele Spitäler nicht über umfangreiche IT-Ressourcen verfügen und aufgrund von Datenschutzanforderungen nicht ohne Weiteres auf Cloud-Dienste zugreifen können. Unser Ziel war es daher, ein performantes Modell für die automatisierte Codierung von medizinischen Textdaten zu entwickeln, das lokal und mit begrenzten Ressourcen einsetzbar ist.
Die daraus resultierte Forschungsarbeit wurde als wissenschaftliche Studie «Evaluating Fine-Tuned Small Language Models for ICD-10 Diagnosis Coding» von Victor Palacios, Curdin Marxer, B. Sunshine und Prof. Dr. Yves Staudt auf der Swiss Data Science Conference 2025 in Zürich vorgestellt und veröffentlicht.
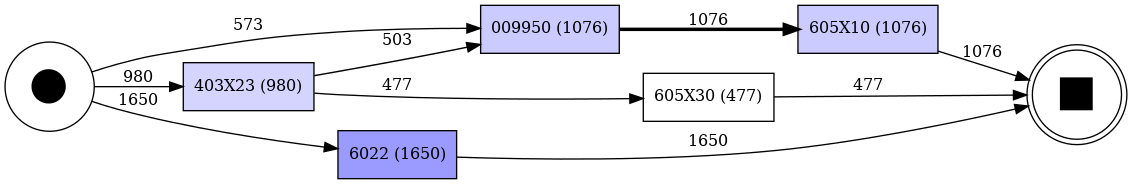
Nachdem die automatische Verknüpfung von Text und medizinischen Codierungen erfolgreich umgesetzt werden konnte, wurden in einem weiteren Schritt die Behandlungspfade mithilfe statistischer und prozessorientierter Methoden, zusammengefasst unter dem Fachbegriff «Process Mining», analysiert und visualisiert. Eine solche Darstellung wurde exemplarisch für Prostatakrebspatienten auf Basis der Gesundheitsversorgungsstatistik 1998–2022 des Bundesamts für Statistik erstellt (siehe Abbildung 1).
Aktuell wird an einer Implementierung unseres Ansatzes auf Daten des Tumorzentrums des Kantonsspitals Graubünden gearbeitet. In einem nächsten Schritt soll dieser in eine interaktive Visualisierung mittels eines Dashboards überführt werden, um dem medizinischen Fachpersonal eine intuitive und praxisnahe Nutzung zu ermöglichen. Unser besonderer Dank gilt dem Förderverein der Fachhochschule Graubünden sowie der Fachhochschule Graubünden für die finanzielle Unterstützung dieses Forschungsprojekts und die Möglichkeit.
Resultate
Als Resultat des Projekts ist folgender wissenschaftlichen Artikel entstanden: «Evaluating Fine-Tuned Small Language Models for ICD-10 Diagnosis Coding» von Victor Palacios, Curdin Marxer, B. Sunshine und Prof. Dr. Yves Staudt
Team
Das Projekt wurde ausserdem von folgenden Personen unterstützt:
- Prof. Dr. med. Roger von Moos - Direktor Tumor- und Forschungszentrum FMH Med. Onkologie, Innere Medizin – Kantonsspital Graubünden
- Lucas Basler – Senior Radiation Oncologist & Data Scientist / Business Analyst & Project Manager - Artificial Intelligence (AI) – Kantonsspital Graubünden
- Rosaria Tino-Corrado – Assistentin Tumor- und Forschungszentrum – Kantonsspital Graubünden
- Prof Dr. Joël Wagner – Full Professor at University of Lausanne (HEC) | Director of the Department of Actuarial Science | Member of the Board of Directors of Retraites Populaires and LALUX Assurances
- Selina Steiner – Praktikantin bei Prof. Dr. Yves Staudt
- Raphael Brunold – Praktikant bei Prof. Dr. Yves Staudt
- Victor Palcios – University of San Francisco, Director Data Science Partnership
Weiterführende Information
Beteiligte
Das Projekt wurde vom Institut für Data Analysis, Artificial Intelligence, Visualization und Simulation (DAViS) im Auftrag des Kantonsspitals Graubünden umgesetzt.